近日,湖北大学生命科学学院、省部共建生物催化与酶工程国家重点实验室谈晓明教授开发了新型蓝藻组学数据库CyanoOmicsDB (http://www.cyanoomics.cn/),相关研究成果以“CyanoOmicsDB: an integrated omics database for functional genomic analysis of cyanobacteria”(《CyanoOmicsDB:一个用于蓝藻功能基因组分析的多组学整合数据库》)为题,在线发表于Nucleic Acids Research。生命科学学院培养的第一届生物信息学专业本科毕业生周鹏为论文共同第一作者,谈晓明教授为通讯作者。
蓝藻(又称蓝细菌)是一类能够进行产氧型光合作用的原核微生物,是地球淡水和海洋生态环境中重要的初级生产力,在地球碳氮等物质循环中具有举足轻重的作用。由于具有较快的生长速度和相对成熟的遗传操作系统,蓝藻一直是光合作用、生物固氮和昼夜节律等基础研究的重要对象。近年来,随着代谢工程和合成生物学研究的兴起,蓝藻又被发展成为一类重要的光合微生物底盘。
随着高通量测序技术的成熟,公共数据库已积累了约1000种蓝藻的基因组序列(全基因组序列或草图),以及丰富的转录组学和蛋白质组学数据。然而,这些分散存储在不同数据库的大量组学数据尚未得到系统整合与挖掘。
为此,谈晓明教授团队收集并整理了来自NCBI Assembly、GEO、SRA数据库和文献的蓝藻多组学数据,最终设计构建了新型的蓝藻组学数据库CyanoOmicsDB(图1)。该数据库收录来自928个蓝藻基因组的8335261个基因条目,并为每一个基因提供多种基因标识符、基因功能注释、参考文献、与其他功能数据库的交叉引用信息以及可视化的基因组位置信息。针对代表性的模式蓝藻,该数据库还提供了经质谱鉴定的肽段、蛋白质翻译后修饰信息、转录起始位点以及多种环境条件和遗传背景下基因差异表达信息。该数据库网站为本领域研究人员提供了便捷的基因功能信息检索服务,将助力蓝藻基因功能相关的科学研究。
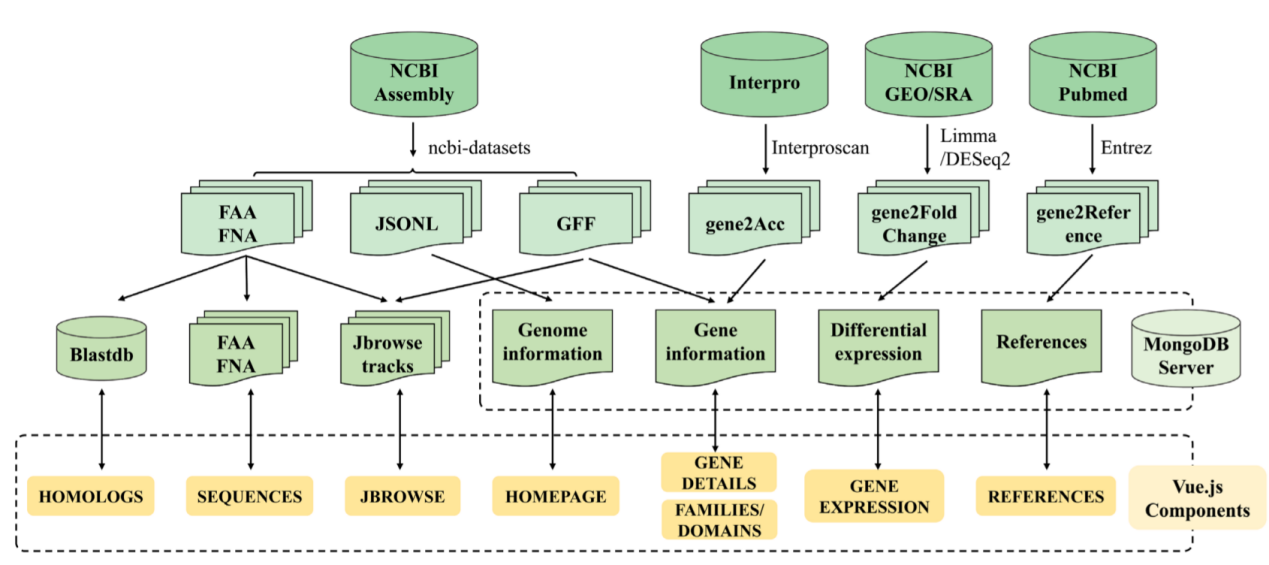
图1 CyanoOmicsDB数据库创建流程图
谈晓明教授一直从事蓝藻遗传学和合成生物学研究,研究成果发表于MetabolicEngineering、Applied and Environmental Microbiology和Biotechnologyfor Biofuels等专业学术期刊,相关研究工作得到了国家自然科学基金、国家重点实验室等多项科研项目的支持。
论文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab891/6382383
(审稿:谢玉平)